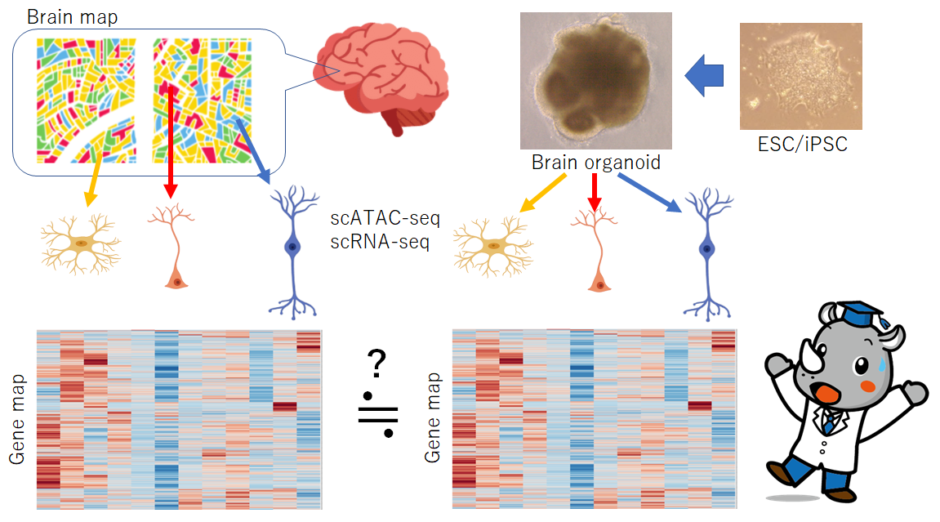
最近、ヒト脳の仕事が立て続けに出ていて迷ってしまいますが、
今日は、ヒト脳の細胞核クロマチン構造の変化を調べた論文です。
scATAC-seqを使って、細胞核の状態を調べた論文から、
前回のレチノイン酸シグナルに話がつながるのが驚きです。
“Single-cell epigenomics reveals mechanisms of human cortical development”
Ryan S Ziffra et al., Nature. 2021 Oct;598(7879):205-213. doi: 10.1038/s41586-021-03209-8. Epub 2021 Oct 6.
https://www.nature.com/articles/s41586-021-03209-8
・ヒト脳のいくつかの部分から細胞を1つ1つ取り出し、クロマチン構造を解析した。
・細胞の種類に特異的な遺伝子発現制御領域を特定した。
・特定された制御領域と神経発達障害、精神疾患に関連領域との相関が見られた。
・細胞種類ごとのクロマチン構造変化と遺伝子発現データの対応づけができた。
・脳領域の特異性にレチノイン酸シグナルが関連していることがわかった。
・脳オルガノイドを用いて脳の発生をクロマチン構造で評価できることを裏付けた。
今回の論文ですが、
この分野の基礎知識が無い人には少し難しいかもしれません。
なるべく分かりやすく解説していきますね。
前提として、エピジェネティクスについて、
細胞の中ある核には我々の遺伝子がコードされている染色体があり、
これはDNA(デオキシリボ核酸)で構成されています。
DNAはヒストン(タンパク質)で巻かれており、クロマチン構造を作っていますが、
このヒストンが色々修飾されることで、クロマチン構造が凝集したり、緩んだりして、
遺伝子の発現が制御されていることが知られています。
例えばMBLサイトがわかりやすい↓
https://ruo.mbl.co.jp/bio/product/epigenome/article/histone-modification.html
これによって、DNA配列は同じであっても、
様々な種類の細胞が分化してくると言うわけです。
今回出てくる、scATAC-seqについて、、、の前にATAC seqについて。
これを細かく話すとキリが無いのですが、ざっくり言うと、
クロマチンのオープンな部分を狙って、前にも出てきたトランスポゾンのシステムを使うことで、
DNAの領域をタグ標識をします。
これを次世代シークエンサーで、読んでやることでどこの部分がオープンか分かるわけです。
Activemotifサイトが詳しい↓
https://www.activemotif.jp/blog-atac-seq
最近では、技術革新が起こって、一つの細胞で解析ができるようになりました。
これで、Single cell (sc) ATAC-seqという訳です。
<論文の流れ>
・脳の発生時のクロマチン状態を探る
各脳領域、dorsolateral prefrontal cortex (PFC),
primary visual cortex (V1), primary motor cortex (M1),
primary somatosensory cortex, dorsolateral parietal cortex,
temporal cortex, insular cortex, the medial ganglionic eminence (MGE)
から、サンプルして、scATAC-seqで、1細胞のエピゲノムを調べた。
クロマチンがオープンな領域から遺伝子活性を推定
細胞の種類をクロマチン状態から判別することができた。
・細胞特異的なエンハンサー領域を特定する
各細胞群に特異的にオープンになっている領域を特定できた。
これらはエンハンサー領域が含まれていると考えられ、さらに
ヒストンH3の27リジンのアセチル化(H3K27ac)による解析
染色体の3次元構造に依存した(Hi-C)解析、
遺伝子発現データを組み合わせることで、
25,659箇所のエンハンサーと推定される領域を同定した。
また、これらの領域のなかで、
ヒストンH3の4リジンのトリメチル化(H3K4me3)による解析
を行うことで、プロモーターと相互作用をしていると考えられる
10,050箇所のエンハンサー領域をさらに絞り込んだ。
細胞に特異的な転写因子の結合サイトと、
これらの細胞特異的なエンハンサー領域は、相関していた。
・神経疾患に関わる制御領域の探索
神経発達障害(NDD)や精神疾患(統合失調症、自閉スペクトラム症ASDなど)は、
特定の遺伝子変異で説明できないことが多く、
ゲノムをコードしない領域や、未知のクロマチン制御因子の変異ではないか、
ということが示唆されている。
そこで、今回特定された、制御領域とこれらの疾患との関連を調べた。
発達遅滞と相関のある遺伝子コピー数変異との関連を見たところ、
deep layer (cortical layers V–VI) excitatory neurons (dlENs)
endothelial/mural, microglia に特異的な、ATACseqピークで見られた。
一方で、Interneuronのプロモーターに関連したピークでも関連が見られた。
ただ、個々の制御領域の特異性は見られなかった。
自閉スペクトラム症(ASD)と神経発達障害(NDD)に関連した領域と
今回得られたピークとの相関を見ると、ほとんどの細胞で関連が示唆された。
また、Common variants(頻度の高い変異)で、
精神疾患のリスクと特定された領域との関連を見ると、
興奮性神経、抑制性神経に特異的なピークが特に関連があった。
・神経発生時のクロマチン状態の動的変化
Visual CortexのscRNA-seqとscATAC-seqのデータセットを使って、
比較解析している。この時、Pseudotime(疑似時間的)解析を使うことで、
放射状グリア(radial glia)からどのように発生が進んだかを推定した。
結果、25,000peaksから、5,000の推定エンハンサー領域を特定し、
これらの中で、遺伝子の発現制御領域に近いものは、
その遺伝子の発現に高く相関していた。
これらの結果は神経発生時のクロマチン構造のダイナミクスを示している。
・脳領域特異的なクロマチン状態
前のセクションのPseudotime解析を用いて、二つの脳領域PFCとV1の状態を調べ、
興奮性神経のクロマチン構造の状態が大きく分かれるポイントを見つけた。
この分岐において、どの転写因子の結合配列がオープンになっているか調べると、
POU3F2, MEIS1, TBR1, NEUROD1, NEUROG2, TBX21、
また レチノイン酸 (RA) シグナルのRXR, RAR, TGIF1がPFCで見つかった。
・レチノイン酸が脳皮質の領域を特定している?
さて、iPS細胞を使って、脳構造をつくるオルガノイド技術を使って、
ヒトの発生を模擬する実験ができるようになっていますが、
この技術を使って、前のセクションのRAの仮説を検証しています。
ここでは、初期の脳オルガノイドに、RAの前駆体のVitaminAを添加した群、
加えない群、RA合成阻害剤(DEAB)を加えた群、の3群で比較しています。
分化開始から10週間後、細胞を一つ一つバラバラにして、
scRNA-seqを行ったところ、前脳の興奮神経マーカーFOXG1/NEUROD2,
SATB2, AUTS2が、Vitamin A存在下で多くなっており、
VitaminAなしでは、NR2F1陽性細胞が多くなっていた。
これによりRAシグナルが、脳領域の特定に関与していることが示唆された。
・脳オルガノイドのベンチマーク
この脳オルガノイドのモデルの性質をクロマチン構造から明らかにするために、
健常人から樹立したiPS細胞とES細胞を用いて脳オルガノイドを作製し、
scATAC-seqデータとscRNA-seqのデータを抽出し、このデータを比較した。
前のセクションで用いた遺伝子活性スコアを指標にすると、
得られた細胞は、RG、IPC、抑制性神経、興奮性神経など、
主要な細胞に分類することができたが、脳のサンプルより種類が少なかった。
scATAC-seqデータを解析すると、オルガノイドで見られなかった細胞は、
マイクログリア、血管内皮細胞、アストロサイト、オリゴデンドロサイト前駆細胞で
(これらの細胞は脳オルガノイドでは分化できない種類)
その他に、V1の興奮性神経のピークも見られなかった。
一方で、脳で特定された80%以上のエンハンサー領域がオルガノイドでも特定された。
脳で特定されなかった、領域を調べると、HIF1A, HIF1B, p53があり、
これらは、細胞のストレスに関連する遺伝子群で、
培養系では、脳組織に比べてやはりストレスがかかってしまっていることを示唆している。
・ASD、NDDの原因って特定がやはり難しい
>精神疾患の遺伝子って特定が色々なところで試みられていますが、あまりハッキリしませんね。家族性とは限らなくて環境因子も大きいみたいです。
・scATACseqの方がscRNAseqより領域特異性が出やすい
>遺伝子発現は、クロマチン構造変化の結果と言えるため、ATAC-seqの方が違いが見えやすいのかもしれませんね。
・こちらでもレチノイン酸シグナルが出てきた!
>正直このデータだけだと、レチノイン酸シグナルって主張するのは弱かったんじゃないかなと想像します。その意味で、前回紹介の論文とセットになっているのではないでしょうか?一緒の時期にかぶせてくるのは編集者(ひょっとしたら著者同士も知り合い)も考えていますね。(ちゃんと引用もしています)
・やっぱり脳オルガノイドも使っている!
>前回の時に、脳オルガノイドはやらないのかな?ってコメントしたと思いますが、こちらでやっていることで相補的になっていますね。
哺乳類の発育中、クロマチン状態の違いは細胞分化と一致し、遺伝子調節の状況の変化を反映しています。発達中の脳では、細胞の運命の特定と地形の同一性は、細胞の同一性を定義し、神経発達障害に選択的な脆弱性を与えるために重要です。ここでは、発達中のヒト脳における細胞型特異的なクロマチンアクセシビリティパターンを特定するために、ヒト前脳からの初代組織サンプルにおけるシーケンシング(scATAC-seq)によるトランスポザーゼアクセシビリティの単一細胞アッセイを使用しました。偏りのない分析を適用して、神経新生中にアクセス可能性の広範な細胞型および脳領域固有の変化を受けるゲノム遺伝子座を特定し、統合分析を適用して、細胞型固有の候補調節要素を予測しました。脳オルガノイドは、ほとんどの推定細胞型特異的エンハンサーアクセシビリティパターンを再現しますが、invivoで見られる多くの細胞型特異的オープンクロマチン領域を欠いていることがわかりました。脳領域全体のクロマチンアクセシビリティの体系的な比較により、大脳皮質の神経前駆細胞間の予期しない多様性と、前頭前野の神経系統の同一性の特定におけるレチノイン酸シグナル伝達の関与が明らかになりました。一緒に、私たちの結果は、細胞型の多様性と細胞の運命の仕様の新たなパターンへのクロマチン状態の重要な貢献を明らかにし、皮質発達のモデルとして脳オルガノイドの忠実度と堅牢性を評価するための青写真を提供します。
Ryan S Ziffra et al., Nature. 2021